Autore: Dott. Caterina Durante, Marina Cocchi, Simone Pellacani (Università degli Studi di Modena e Reggio Emilia), Monica Casale, Eleonora Mustorgi (Università di Genova)
Al giorno d’oggi, nel mondo della ricerca scientifica, la Scienza dei Dati, Data Science, ha raggiunto un ruolo prioritario nell’estrarre informazione da set di dati di varia natura, sempre più complessi, ma allo stesso tempo sempre più ricchi di informazione. Nel presente articolo vengono illustrati due approcci scientifici che, combinando insieme le potenzialità dell’analisi dei dati e dei metodi spettroscopici, hanno il principale obiettivo di supportare la ricerca medica nello sviluppo di metodi analitici rapidi e accurati che consentano uno screening precoce del tumore alla prostata direttamente attraverso l’analisi delle urine, al fine di fornire risultati attendibili e di migliorare al contempo la compliance del paziente.
La Scienza dei Dati, Data Science, è l’insieme di metodi scientifici e di analisi dei dati che vengono utilizzati per estrarre informazioni utili al raggiungimento degli obiettivi di una ricerca scientifica. A oggi questa nuova scienza ha assunto un ruolo fondamentale, basti pensare agli ingenti quantitativi di dati (big data) che, quotidianamente, si ottengono in un laboratorio di ricerca grazie all’utilizzo di tecnologie innovative, ma che, a causa del loro volume, possono essere analizzati solo attraverso algoritmi matematici dedicati.
In medicina i big data sono sicuramente generati dalle cartelle cliniche elettroniche, che contengono informazioni sulla storia medica di un paziente (precedenti malattie, terapie, ecc.), ma anche nei laboratori di ricerca, dove i diversi strumenti analitici, che sono in grado di caratterizzare chimicamente un campione (sangue, urina, tessuto, ecc.) in tempi ristretti, producono dei segnali strumentali che rappresentano una vera e propria impronta digitale (fingerprint) del campione analizzato. Questi segnali, abbinati a tecniche di elaborazione dati, possono diventare degli strumenti analitici in grado di supportare la ricerca medica in diversi contesti.
In questo articolo verranno descritti due approcci analitici, basati su tecniche di fingerprint, sviluppati a supporto della diagnosi del cancro alla prostata.
Il cancro alla prostata è il secondo tumore maligno più diffuso nella popolazione maschile, dopo il tumore ai polmoni. L’incidenza e la mortalità del carcinoma prostatico sono fortemente correlate all’aumentare dell’età. Lo sviluppo di questa patologia risulta infatti più comune dopo i 65 anni.
Nella maggioranza dei casi, le fasi iniziali di questa neoplasia risultano del tutto asintomatiche rendendo difficile una diagnosi precoce. A oggi, il protocollo per la diagnosi del carcinoma prostatico prevede visita urologica che comporta esplorazione rettale e conseguente dosaggio dell’Antigene Prostatico Specifico (PSA) con un prelievo del sangue. Alterazioni dei normali livelli di PSA possono essere dovuti a molteplici cause, sia fisiologiche sia patologiche, rendendo difficile un’interpretazione univoca dei risultati. L’unico esame in grado di identificare con certezza la presenza di cellule tumorali nel tessuto prostatico è la biopsia prostatica. La mancanza di specificità del dosaggio del PSA e la prospettiva di prevenire biopsie prostatiche invasive e talvolta inutili, suggeriscono la necessità di individuare nuovi metodi di analisi meno invasivi e più specifici. In particolare, sulla base delle più recenti evidenze scientifiche, risulta di attuale interesse disporre di metodi analitici rapidi ed accurati che consentano uno screening precoce del tumore alla prostata, direttamente attraverso l’analisi delle urine, al fine di fornire risultati attendibili e di migliorare al contempo la compliance del paziente. A tale scopo metodi spettroscopici di fingerprint non distruttivi quali la Spettroscopia nel Vicino Infrarosso (NIR) e la Spettroscopia di Fluorescenza, accoppiati ad approcci statistici multivariati per l’interpretazione di segnali spettrali complessi, si sono rivelati estremamente efficienti per l’analisi di fluidi biologici, tra cui l’urina, in ambito clinico [1].
La spettroscopia
La spettroscopia è la scienza che studia l’interazione tra la radiazione elettromagnetica e la materia. Lo spettro della radiazione elettromagnetica è suddivisibile in diversi intervalli di lunghezze d’onda (Figura 1), le cosiddette regioni spettrali, molte delle quali sono di interesse analitico.
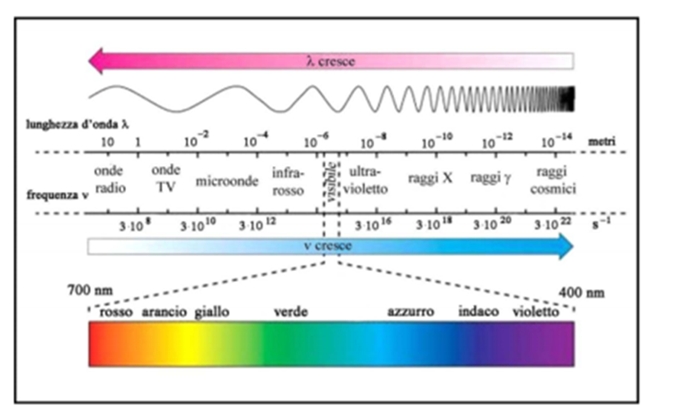
La Spettroscopia, che sfrutta la regione dell’infrarosso, viene definita spettroscopia vibrazionale. Gli atomi legati fra di loro da legami covalenti non sono fissi, ma sono in continua vibrazione. Questo significa che la lunghezza e gli angoli dei legami cambiano continuamente per via di queste vibrazioni, che a temperatura ambiente sono di pochi nanometri. Quando una molecola assorbe la radiazione infrarossa si ha un aumento (inteso come aumento dell’ampiezza della vibrazione) della vibrazione dei legami e produce dei segnali che sono tipici dei legami coinvolti. In particolare, le bande di assorbimento più importanti che si incontrano nella regione del Vicino Infrarosso (NIR) sono dovute ai gruppi funzionali dei legami O-H (ad esempio acqua, grassi o carboidrati), legami C-H (tutti i composti organici in generale) e legami N-H e S-H (ad esempio proteine e amminoacidi).
La spettroscopia di fluorescenza è una tecnica di emissione ottica rapida e sensibile in cui, eccitando le molecole di un campione attraverso una sorgente di fotoni, è possibile misurare l’intensità della radiazione elettromagnetica che esse emettono passando dallo stato eccitato a quello fondamentale.
I campi di applicazione sono i più disparati: dalla diagnostica ambientale, industriale e medica, sino al sequenziamento del DNA, all’analisi genetica e alle biotecnologie. In particolare, una matrice di emissione ed eccitazione, EEM, è una matrice tridimensionale a cubo (Figura 2) che contiene i dati sui campioni rapportati alle lunghezze d’onda di eccitazione e di emissione. In medicina, questa tecnica è stata impiegata sin dagli anni Ottanta per l’analisi dei fluorofori contenuti nel plasma di pazienti affetti dal cancro. Ciascun fluoroforo emette una fluorescenza caratteristica, per identificare il tipo di molecola è sufficiente osservare la posizione del picco nella regione dello spettro di eccitazione-emissione. L’intensità del picco, invece, ci consente di determinare la concentrazione del fluoroforo nel campione.

Entrambe le tecniche presentano innumerevoli vantaggi che rendono ragione del loro notevole sviluppo e campo di applicazione, tra i quali: (i) l’acquisizione degli spettri relativi a campioni solidi e liquidi, (ii) delle tempistiche molto brevi, (iii) nessun tipo di pretrattamento del campione sottoposto ad analisi, e (iv) sono delle tecniche economiche e non inquinanti (green chemistry).
Sia gli spettri NIR che le matrici EEM sono segnali complessi, spesso caratterizzati da bande larghe e sovrapposte. L’estrazione dell’informazione chimica utile contenuta in questi segnali non può essere ricavata da un’interpretazione diretta dello spettro, ma richiede l’impiego di metodi statistici multivariati che vanno sotto il nome di chemiometria.
La chemiometria è una disciplina che, tramite metodi matematici e statistici, viene utilizzata per approfondire le conoscenze su sistemi chimici, fornire il maggior numero possibile di informazioni chimiche rilevanti analizzando i dati chimici (Figura 3) e progettare o selezionare procedure sperimentali ottimali.
In questo contesto risultano di fondamentale importanza i metodi esplorativi “unsupervised”, che consentono di selezionare variabili chimiche rilevanti ed eliminare quelle ’rumorose’, di trovare aspetti comuni tra campioni e rilevare gruppi e tendenze nei dati. Tra le tecniche esplorative quella più importante è sicuramente l’Analisi delle Componenti Principali (PCA) [2] perchè consente di ridurre la dimensionalità di una matrice complessa di dati e permette di migliorare l’interpretazione dei risultati migliorandone la visualizzazione mediante l’utilizzo di grafici. In particolare, il grafico degli scores (le coordinate dei campioni nello spazio delle componenti principali) consente di visualizzare eventuali raggruppamenti o tendenze dei campioni. Il grafico dei loadings (i coefficienti delle combinazioni lineari) permette di ottenere informazioni su come le variabili sperimentali sono correlate tra di loro e su come queste influenzano la distribuzione dei campioni nel grafico degli scores.
Un’altra tecnica che sta acquisendo sempre più interesse nell’ambito chemiometrico è PARAFAC (Parallel Factor Analysis) [3, 4]. Questa tecnica è in grado analizzare set di dati (dati multidirezionali) caratterizzati da più fonti di informazione. Esempi chimici possono essere gli spettri di emissione di fluorescenza misurati a diverse lunghezze d’onda di eccitazione per diversi campioni. La determinazione di tali variabili darà origine a dati tridimensionali, cioè disposti in un cubo, anziché in una matrice come nei set di dati multivariati standard.
Sviluppo di un protocollo semplice e non invasivo per la diagnosi del cancro alla prostata
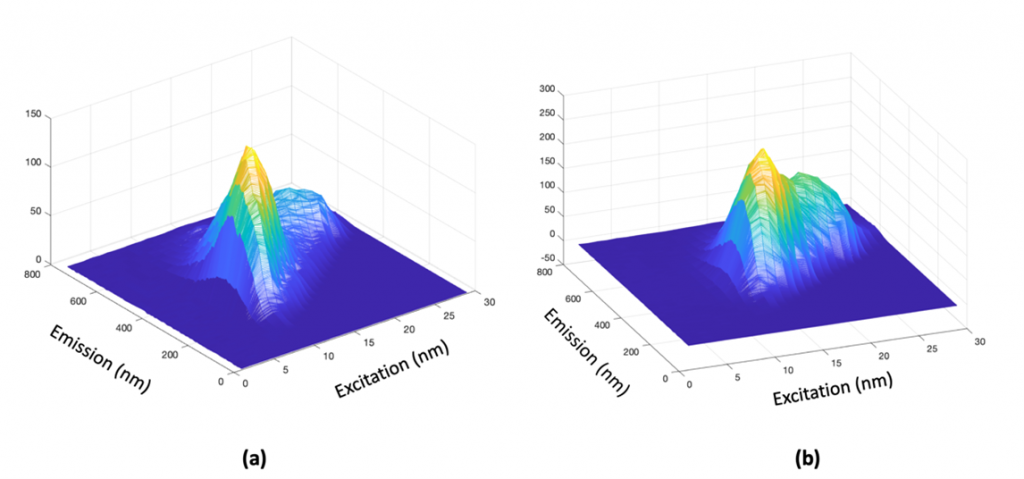
Le tecniche spettroscopiche precedentemente descritte abbinate ad analisi chemiometrica, hanno dimostrato delle notevoli potenzialità nello sviluppo di strumenti in grado di supportare la diagnosi del cancro alla prostata. In particolare, sono state impiegate per lo sviluppo di un protocollo semplice e non invasivo che ha riguardato l’analisi di campioni di urine provenienti da pazienti con carcinoma prostatico istologicamente provato e da donatori sani. Nella Figura 4, sono stati riportati, a titolo esemplificativo, due segnali ottenuti per un paziente malato e per un donatore sano.
I segnali ottenuti sono stati utilizzati come impronta digitale dei campioni investigati e sono stati sottoposti a elaborazione statistica mediante analisi multivariata. Nella Figura 5 è stato riportato il grafico degli scores ottenuto dall’analisi delle componenti principali applicata ai segnali ottenuti.
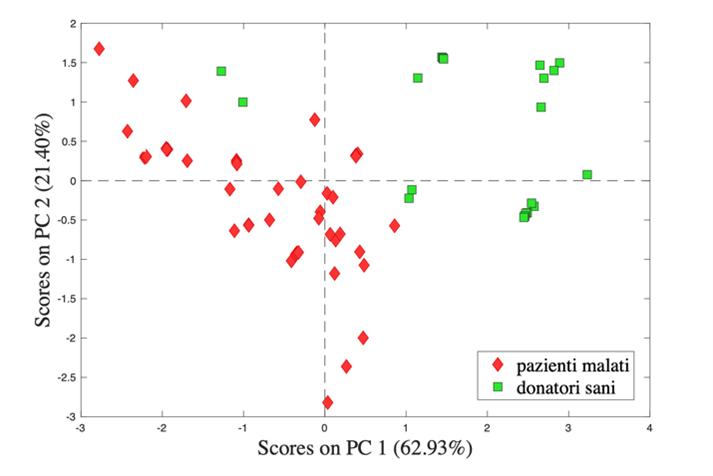
Ogni simbolo rappresenta un campione di urina analizzato (in verde i campioni provenienti dai donatori sani e in rosso quelli provenienti dai pazienti affetti da tumore prostatico) e la posizione di ogni simbolo nel grafico è funzione di tutta l’informazione contenuta nel segnale di fingerprint del campione corrispondente. Dal grafico è evidente che l’utilizzo di questa tecnica permette di differenziare i campioni di urina provenienti dai donatori sani da quelli provenienti dai pazienti; differenze rese possibili dalla presenza di biomarkers che caratterizzano gli spettri di fluorescenza acquisiti. Sicuramente, il numero relativamente basso di campioni analizzati impedisce di poter sviluppare dei modelli di classificazione statisticamente robusti. Tuttavia, i risultati ottenuti rappresentano un ottimo punto di partenza per lo sviluppo di un protocollo semplice e non invasivo per la rilevazione del cancro alla prostata da utilizzare come strumento di screening in grado di supportare i diversi strumenti diagnostici attualmente utilizzati.
Per approfondimenti: (1) Shaw, R. A., & Mantsch, H. H. (2006). Infrared spectroscopy of biological fluids in clinical and diagnostic analysis. Encyclopedia of Analytical Chemistry: Applications, Theory and Instrumentation.; (2) S. Wold, K. Esbensen, P. Geladi. Principal component analysis. Chemometr. Intell. Lab. Syst., 2 (1987), pp. 37-52 ; (3) K.R. Murphy, C.A. Stedmon, D. Graeber, R. Bro, Fluorescence spectroscopy and multi-way techniques. PARAFAC, Anal. Methods 5 (2013) 6557–6566 ; (4) R. Bro, PARAFAC. Tutorial and applications, Chemom. Intell. Lab. Syst. 38 , (1997) 149-171.